近日,信息學院張亮團隊與農學院王宏偉教授在International Journal of Biological Macromolecules上發表了題為“MF-ProtDisMap: protein real-valued distance prediction with fusion of sequence and coevolutionary features”的研究論文。山東農業大學張亮教授和王宏偉教授為該論文的共同通訊作者,山東農業大學碩士張宇菲與廈門大學碩士鐘蘇陽為該論文共同第一作者,山東農業大學為第一通訊單位。
蛋白質結構的精確解析是理解生物微觀分子機制、基因精準設計的基礎。盡管AlphaFold2在蛋白質結構預測方面取得了顯著成果,但如何進一步優化關鍵功能區域(如酶的活性位點、蛋白-配體結合界面)及獨立結構域的預測精度,仍是當前學界關注的核心問題與研究焦點。殘基間實值距離預測不僅為解決上述問題提供了高分辨率的幾何約束,還能直觀反映其內部相互作用與空間組織,從而使模型預測結果具有更強的可解釋性。
團隊基于ESM與MSA Transformer兩個大模型構建了MF-ProtDisMap 大模型框架,該框架通過三項核心設計實現了性能提升:其一,雙通道特征提取模塊同時捕捉蛋白質序列特征與共進化語義信息;
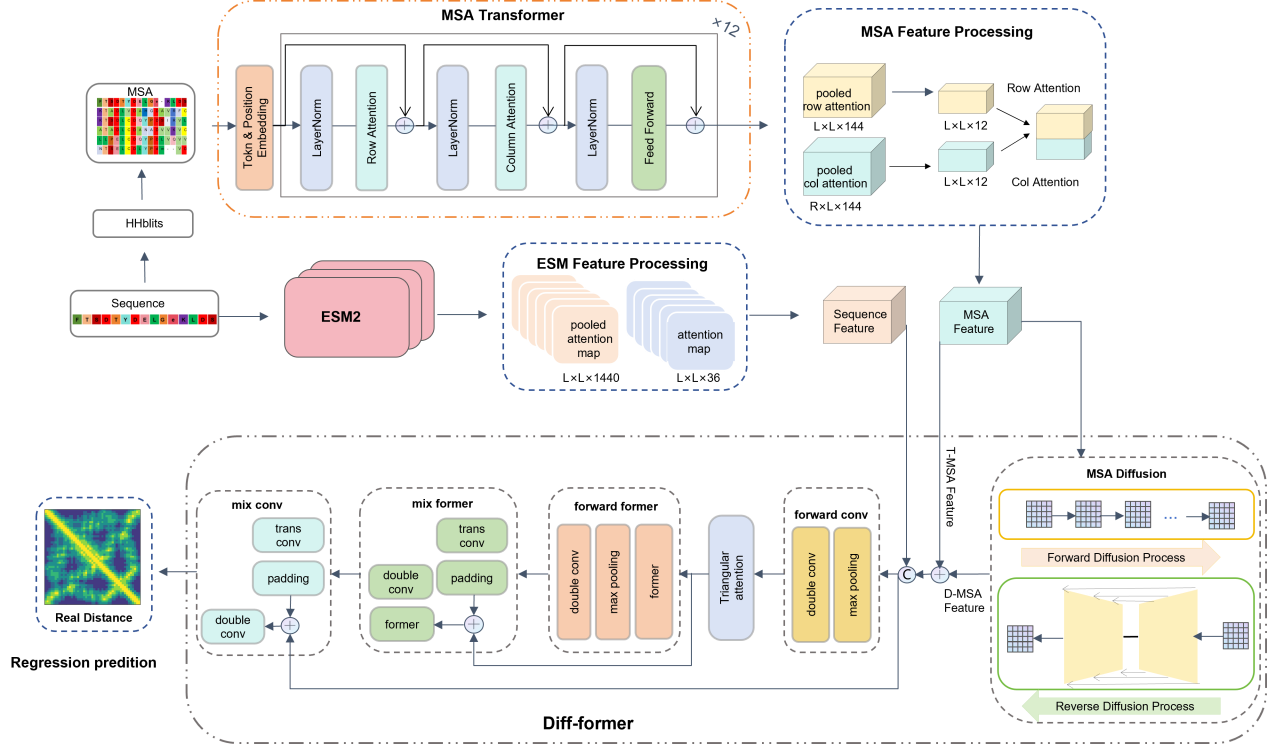
圖1 MF-ProtDisMap 網絡架構
其二,“group pooling”策略實現高效的特征降維與融合;其三,Diff-former 特征學習模塊結合三角注意力機制與擴散模型以強化殘基間關系建模。在蛋白質實值距離預測任務中,MF-ProtDisMap的MAE和RMSE分別為2.20?和3.40?,優于ESMFold的2.56?和5.38?。此外,在接觸預測任務的驗證中,該模型的 ROC 值為 84.56%,PR 值為 81.01%,均超過了現有最佳方法。鑒于缺乏能夠直接從實值距離預測重建蛋白質結構的可用工具,團隊開發了ProtDisFold,用于距離約束結構建模,并成功重建了祖先植物α/β-水解酶(PDB ID:7ukb)、Fhb7-GST和植物NLR Sr35的三級結構。本研究將助力作物改良精準設計、基于冷凍電鏡刻畫微觀結構等領域發展。
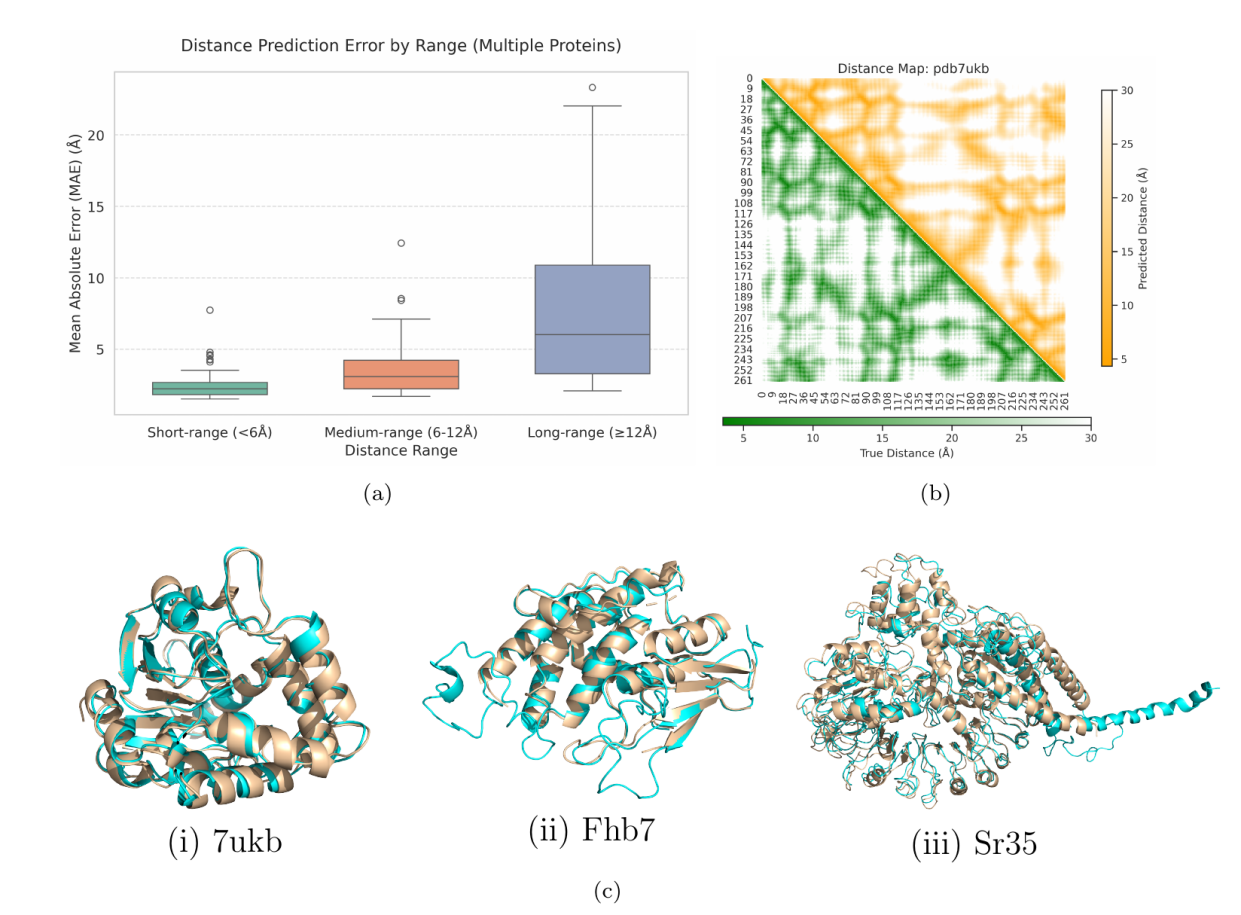
圖2 α/β-水解酶、Fhb7-GST和NLR Sr35的3D結構模型
該研究得到了國家重點研發計劃項目、國家自然科學基金、山東省自然科學基金的資助。
論文鏈接:https://doi.org/10.1016/j.ijbiomac.2025.147637
編 輯:萬 千
審 核:賈 波









